On the journey of
[논문읽기] Brain tumor segmentation based on deep learning and an attention mechanism using MRI multi-modalities brain images 본문
[논문읽기] Brain tumor segmentation based on deep learning and an attention mechanism using MRI multi-modalities brain images
dlrpskdi 2023. 11. 16. 21:09Original Paper) https://pubmed.ncbi.nlm.nih.gov/34035406/ (Brain tumor segmentation based on deep learning and an attention mechanism using MRI multi-modalities brain images)
Brain tumor segmentation based on deep learning and an attention mechanism using MRI multi-modalities brain images - PubMed
Brain tumor localization and segmentation from magnetic resonance imaging (MRI) are hard and important tasks for several applications in the field of medical analysis. As each brain imaging modality gives unique and key details related to each part of the
pubmed.ncbi.nlm.nih.gov
0. Abstract
- 이미지 전체보다는 작은 부분에 작용하는 preprocessing 접근법을 제안하여 유연하고 효과적인 brain tumor segmentation을 얻는다.
- Distance-Wise Attention(DWA) 메커니즘을 제안하여 segmentation 정확도를 높인다.
- 기존의 연구들은 주로 T1, T1c, T2, FLAIR의 4가지 modality들을 사용한 complex structure로 인해 train과 test에 많은 시간이 소요되었다. 이에 비해, 본 논문에서는 간단하고 효율적인 Cascade convolutional neural network(C-CNN)을 제안하여 local과 global feature 모두를 잘 포착하고자 하였다.
- 기존 연구들의 4개 modality에 대한 설명
- T1: T1 가중 영상은 뇌의 일반적인 구조를 보여주는 데 유용하다. 이 기법에서는 뇌의 백질이 어둡고 회백질이 밝게 보임
- T1c 또는 T1 with contrast (T1 강조): 이 기법은 조영제를 사용하여 혈액-뇌 장벽의 무결성을 평가. 조영제는 혈액-뇌 장벽을 통과할 수 없으므로, 조영제가 뇌 조직으로 유출되는 부위는 장애가 있는 것으로 간주되며, 종양이나 염증과 같은 병변을 감지하는 데 도움이 된다.
- T2: T2 가중 영상은 뇌의 수분 함량을 강조하여 보여준다. 이 기법에서는 뇌의 백질이 밝고 회백질이 어둡게 나타나며, 뇌종양, 뇌졸중, 염증 등의 병변을 감지하는 데 유용하다.
- FLAIR (Fluid Attenuated Inversion Recovery): FLAIR 기법은 T2 영상의 한 형태로, 물의 신호를 억제하여 병리학적인 수분(예: 부종)이 더 잘 보이게 하는 기법이다. 이는 특히 다발성 경화증 또는 다른 형태의 뇌 병변을 감지하는 데 도움이 된다.
1. Introduction
자동적이고 robust brain tumor segmentation은 brain tumor의 진단과 치료에 큰 영향을 줄 것이다. Tumor와 NAT(normal adjacent tissue) 사이에는 여러 차이점들이 있고 정확하고 brain tumor의 효과적인 boundary curves를 찾는 몇몇 모델들의 있다. 이는 다음과 같이 세 가지의 큰 카테고리로 분류할 수 있다.
- Hand-crafted features
- Multi-atlas algorithm(MAS)
- Deep learning method
2. Material and Method
1) Pre-processing (전처리)
다른 많은 딥러닝 접근법들과 달리 중요한 feature를 추출하기 위해 이미지 전체가 아닌 제한된 영역에만 집중을 한다.
1. Similar distributions
최종 segmentation 정확도를 높이기 위해 T1, FLAIR, T1C, T2의 네가지 모달리티를 사용한다. MRI 데이터를 더 동일하게 하고 anisotropic을 제거하기 위해 Z-score normalization을 적용하여 평균이 0이 되고 분산이 1이 되게 만든다.
2. Tumor representation

연속된 슬라이스에서 tumor의 위치는 같지만, 점점 증가해서 maximum size를 찍은 후 사라질 때까지 점점 감소한다.
3. Finding the expected area of the tumor


연구 단계에서는 이 tumor가 나타났다가 사라지는 위치를 찾는 것이 어렵지만 biggest tumor를 찾을 때는 그렇지 않다. Tumor area를 찾는 것은 다음과 같은 단계를 따른다.
- T1을 제외한 모든 모달리티들을 읽고 z-score normalization을 계산한다.
- 얻은 이미지에서 threshold를 FLAIR와 T2에는 0.7로, T1ce에는 0.9로 주어 이진화한다.
- 관계없는 영역을 제거하기 위해 morphological operator를 적용한다.
- Fig 4, 5에서 보다시피 tumor-like objects가 tumor인지 아닌지를 결정하기 위해 binarized T1ce images에 추가 제약 조건을 건다.
- Solidity > 0.7
- Area > 500 pixels
- Major axis length > 35 pixels
- Fig 4, 5에서 보다시피 tumor-like objects가 tumor인지 아닌지를 결정하기 위해 binarized T1ce images에 추가 제약 조건을 건다.
- FLAIR과 T2 이진 이미지를 곱해서 새로운 이미지를 만든다.
- 이때 T1ce와 dot product of FLAIR, T2 내부의 binary object가 겹치는 영역이 20 pixels보다 큰 것만 고려한다. (fig 5)
- 각 이미지에서 얻은 영역 들을 결합한다.
마지막으로 큰 tumor의 위치를 찾아야 하는데, 연속된 슬라이스를 확인했을 때 tumor object가 같은 위치에서 조금씩 크기 변화가 있다면 이 object를 tumor로 확신할 수 있다.
과정을 정리하자면 다음과 같이 나타낼 수 있다.

이렇게 object와 그 위치를 찾으면 모든 슬라이스에서 tumor를 찾고 segment하기 위해 이 영역에만 집중 할 수 있다.
2) Deep learning architecture
이 논문에서는 먼저 높은 확률로 tumor가 있을 관심 영역을 찾고, CNN 모델을 그 작은 영역에 적용한다.
CNN의 가장 큰 문제점은 fuzzy segmentation 결과를 가진다는 것과 convolution, pooling operation의 strides로 인해 spatial information이 감소한다는 것이다. 이러한 segmentation의 정확도와 효율을 개선하기 위해 dilated convolution/pooling, skip connection, attention mechanism 등이 제안되었다.
3) Proposed structure
이 논문에서는 cascade CNN 모델을 통해 다른 MRI 모달리티들 간의 local과 global information을 결합하고, distance-wise attention mechanism을 통해 4가지의 입력 모달리티에서의 brain tumor 위치 영향을 고려한다.
Distance-wise attention module(DWA)

Binary mask의 중심과 expected area의 중심의 불일치를 이용해 각 픽셀의 확률을 추측할 수 있는데 이 방법을 이용한 것이 DWA이다.



x_c, y_c를 object의 중심이라고 하고 W_{object}H_{object}를 각각 object의 너비와 높이라고 할 때 이와 같이 distance-wise를 정의할 수 있다.

이 과정은 모든 입력 모달리티에서 수행되고 모두의 평균이 각 슬라이스의 모듈의 output으로 주어진다.

전체적인 프로세스는 아래와 같다

Expected area의 중심이 있는 patches를 추출하는 게 유의미하다.
3. Experiments - BRATS 2018 dataset 사용
1) Data and implementation details
- BRATS 2018 dataset
- 멀티 모달 MRI 이미지와 임상 데이터
- 240 x 240 x 150
- 다양한 자기장 강도, 스캐너
- experiments 조건?
- Intel Core I7- 3.4 GHz, 32 GB RAM, 15 MB Cache, CUDA 9.0, CuDNN 5.1, GPU 1080Ti NVIDIA computer 64bit os에서 MATLAB 사용
- train step
- Adam
- batch size = 2
- weight decay = 10^-5
- initial learning rate = 10^-4
- total : 13h to train, 7 s per volume to test.
2) Evaluation measure
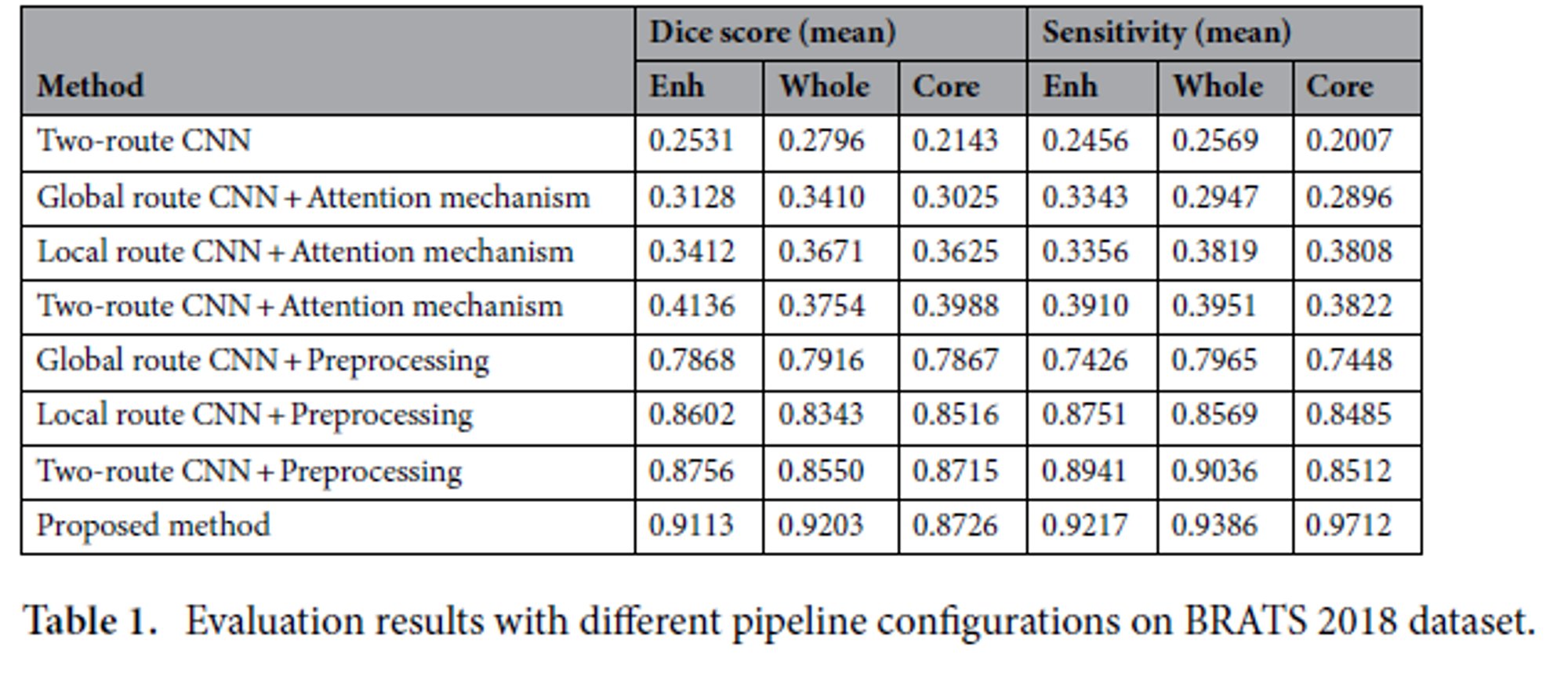

3) Evaluation results
implemented five other models-(Multi-Cascaded, Cascaded random forests, Cross-modality, Task Structure, One-Pass Multi-Task
only Two-route method >> bad
Two-route + attention >> good
Two-route + 전처리 >> very good
전처리를 추가하면 dice 점수가 다 오름

End, Whole, Core 각각에 대해 0.2531, 0.2796, 0.2143에서 0.8756, 0.8550, 0.8715로 증가


- T1C 이미지 내의 tumor core와 enhancing tumor 영역 간의 차이로 인해, 다른 모달리티를 사용하지 않고도 높은 정확도로 사이의 경계를 쉽게 구별할 수 있음.
- 그러나 종양 핵심, 부종 영역, 강화된 부종 영역의 경계를 다룰 때는 사실이 아님.
- 각 모달리티의 특성으로 인해, 검색 영역을 줄이면 deep CNN 모델이 필요하지 않다는 것을 관찰할 수 있다
- DWA 모듈의 사용으로 인해 종양과 뇌에서 독특한 문맥의 정보를 추출할 수 있어, 더 나은 segmentation 얻음.
4. Discussion and conclusions
이 논문에서는 ....
- 4개의 MRI 모달리티의 특성에 따라 새로운 brain tumor segmentation architecture를 개발
- 종양 조직 근처의 뇌 이미지 일부분(전처리해서 20%만 사용)에서만 작업해도 사람이 본 것과 같은 성능 달성 CNN 모델에 효과적인 것을 입증, 시간단축
- 캐스캐이드 CNN 모델로 지역 및 전역 특징을 두 가지 방법으로 추출
- 강력한 전처리 기법을 사용하여 종양의 예상 영역을 추출한 후, 패치들을 네트워크에 보냄
- DWA 매커니즘의 효과와 other method와 비교했을 때 논문에서 제시한 모델이 뛰어난 성능 보여줌
- 뛰어난 모델이지만, 종양이 뇌를 1/3 이상일 경우 한계가 있음.



